新冠Mu(B.1.621)及C.1.2突变株抗原产品现货发售
为助力针对新冠病毒Mu(B.1.621)和C.1.2突变株的相关研究,ACRO第一时间快速响应,全力加速相关抗原蛋白的开发,现已成功表达出Mu(B.1.621)和C.1.2突变株的Spike,RBD和NTD抗原蛋白并完成结合活性验证,可用于新冠疫苗对突变株保护效果及新冠检测试剂检出率的相关研究。
欢迎联系文末小助手,订购现货抗原产品!























新冠Mu(B.1.621)及C.1.2突变株抗原产品现货发售
为助力针对新冠病毒Mu(B.1.621)和C.1.2突变株的相关研究,ACRO第一时间快速响应,全力加速相关抗原蛋白的开发,现已成功表达出Mu(B.1.621)和C.1.2突变株的Spike,RBD和NTD抗原蛋白并完成结合活性验证,可用于新冠疫苗对突变株保护效果及新冠检测试剂检出率的相关研究。
欢迎联系文末小助手,订购现货抗原产品!
自新冠疫情爆发以来,世界各地的科学家正在以惊人的速度开发和生产疫苗以阻止 COVID-19疫情的传播;试剂厂商同样全力以赴生产制备新冠检测试剂已保证准确高效的检出新冠感染病例。
然而,不断出现的变异毒株使得抗疫形势依旧严峻。9月23日,美国FDA紧急通知,要求所有在之前获得EUA授权的新冠检测试剂制造商增加新冠检测试剂针对不同病毒变异的检测功效;疫苗在上市前的临床实验中同样要求对其针对突变株的保护效力进行检测。因此,对于新冠变异毒株的相关研究刻不容缓。
Mu突变株(PANGO谱系编号为B.1.621)于2021年1月在哥伦比亚被首次发现,因其在哥伦比亚和厄瓜多尔等地快速传播并已成为当地的主流毒株,且美国、日本以及欧洲和拉美部分国家均已报告发现Mu变异毒株感染的病例,WHO于2021年8月30日将Mu突变株列为“待观察的突变株”(variant of interest, VOI)。
根据“全球共享流感数据倡议组织”(GISAID)发布的数据,截止8月底,Mu(B.1.621)突变株已经蔓延到40个国家。流行率最高的地区是该毒株的发现地哥伦比亚,目前该毒株已占哥伦比亚所有新冠感染病例的80% [1],此外该毒株在厄瓜多尔、哥斯达黎加等地造成的感染病例数也居高不下,曾引发该毒株是否会替代Delta突变株成为新冠主流毒株的讨论(图1)。
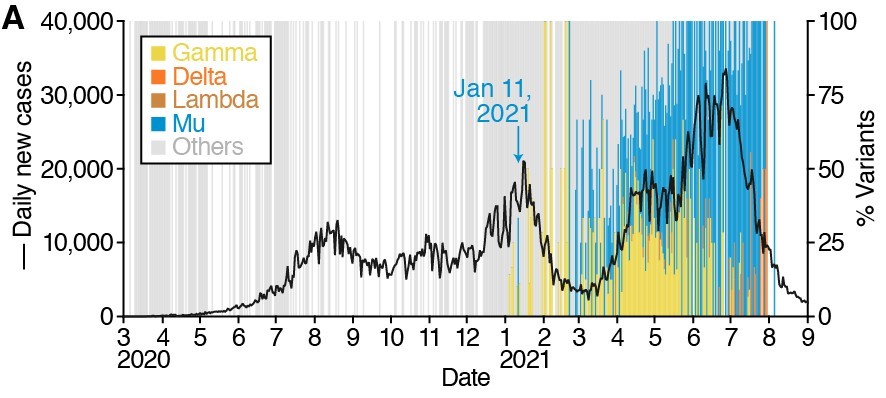
图1. Mu(B.1.621)在哥伦比亚的流行率
根据基因测序结果,Mu(B.1.621)突变株的Spike蛋白上有9个典型突变,其中包括NTD上的T95I/Y144S/Y145N,RBD上的R346K/E484K/N501Y,位于CTD上的D614G, 位于弗林酶切位点附近的P681H以及S2上的D950N [2]。其中E484K/N501Y突变同样存在于Beta和Gamma等具有较强免疫逃逸能力的突变株中,预示着Mu(B.1.621)也可能会导致疫苗的保护效价降低。日本东京大学研究团队的病毒中和实验结果显示[1],康复者血清和辉瑞疫苗接种者血清对Mu(B.1.621)突变株的中和抗体滴度低于Beta突变株,在所有VOC和VOI中导致血清保护抗体滴度下降最多(图2)。
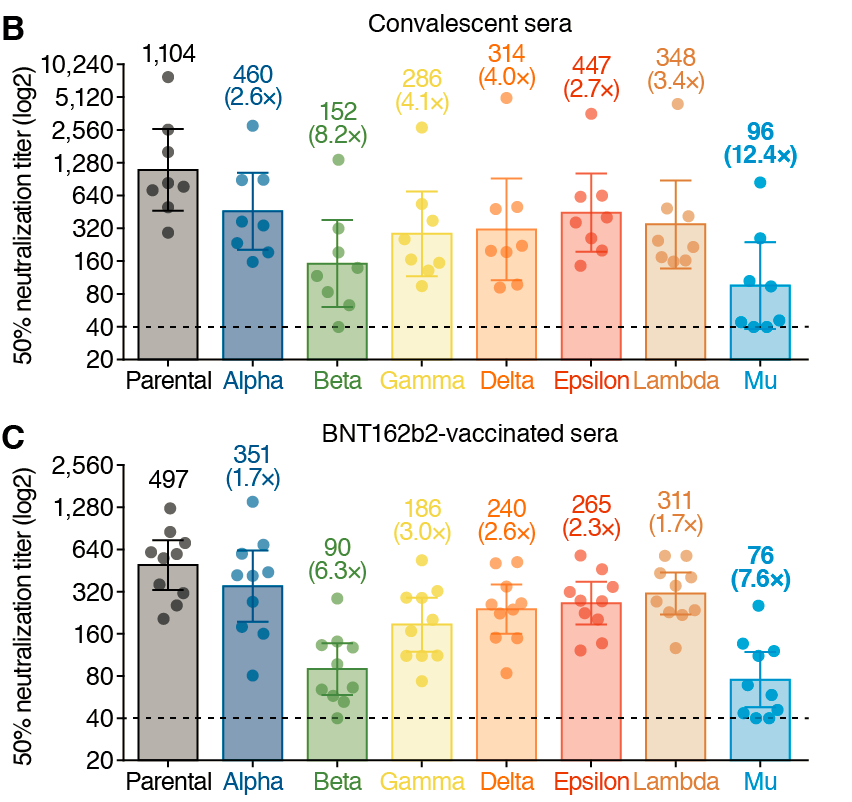
图2. B.1.621(μ)的血清中和测试结果
上述结果表明,Mu(B.1.621)突变株不仅具有极快的传播能力,同时还具有极强的免疫逃逸能力,需要引起政府部门和科研机构的警惕。
C.1.2突变毒株
继出现具有较强免疫逃逸能力的新冠病毒Beta(B.1.351)突变株后,南非又于2021年5月第三波新冠疫情中发现C.1.2突变毒株,目前该突变株已在欧洲、亚洲、非洲和大洋洲的7个国家出现。基因组测序数据显示,C.1.2突变株在南非呈现出与Beta突变株相仿的传播趋势,目前其占比正在逐月增长[3]。
在C.1.2毒株的Spike蛋白序列中出现了14个典型突变[3],其中包括NTD上的5个突变(C136F, Y144del, R190S, D215G, LA242-243del),RBD上的3个突变(Y449H, E484K, N501Y),弗林酶切位点附近的2个突变(N679K, T716I),以及其它突变4个突变(P9L, D614G, H655Y, T859N)。其中E484K/N501Y突变同样存在于Beta和Gamma等具有较强免疫逃逸能力的突变株中,且RBD上的Y449H和弗林酶切位点附近的N679K/T716I突变可能与免疫逃逸及病毒传染性增强相关,预示着C.1.2毒株也可能会导致疫苗的保护效价降低。目前关于C.1.2突变株的相关研究较少,亟需对该毒株的致病性、传染性和免疫逃逸能力进行详细研究。

为助力针对新型冠状病毒变异毒株Mu(B.1.621)和C.1.2的相关研究及疫苗开发,ACROBiosystems已经在第一时间成功研发生产出相应突变株抗原产品。
1
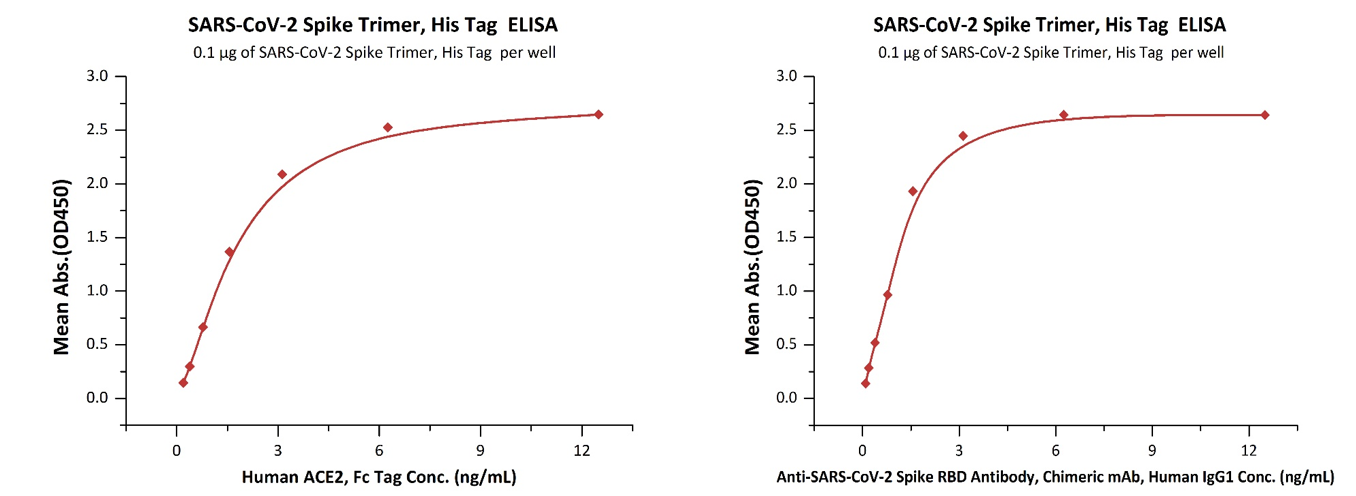
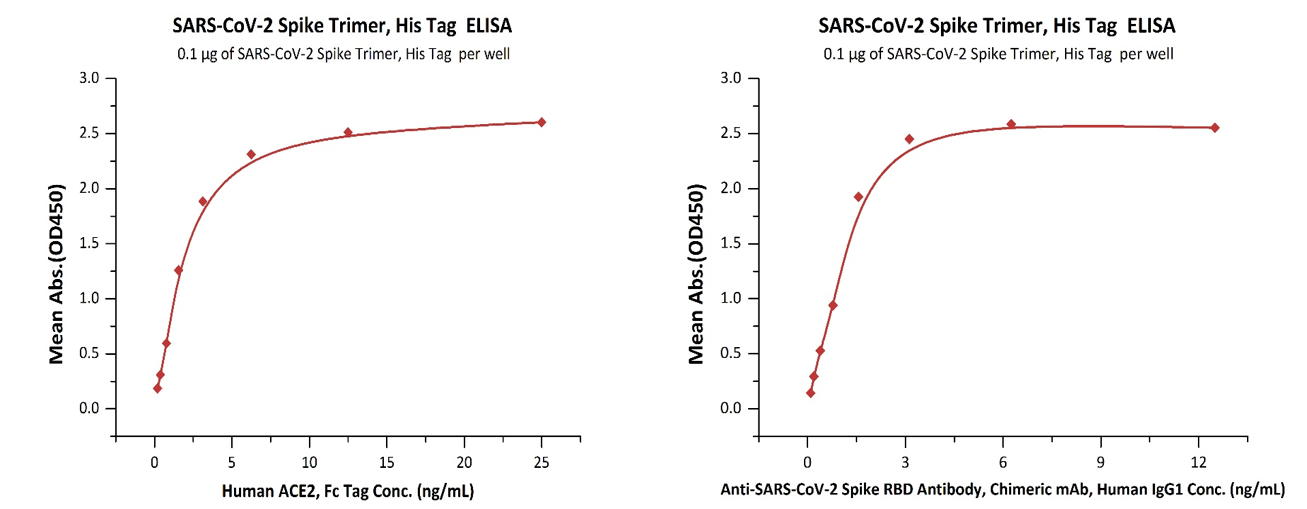
产品包括Spike Trimer, Spike RBD, Spike NTD等;
2
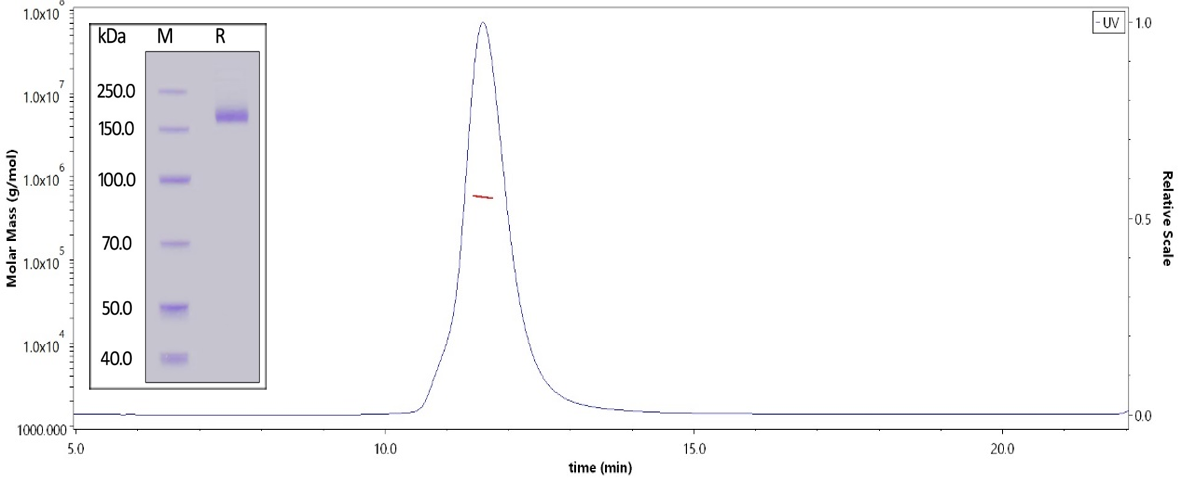
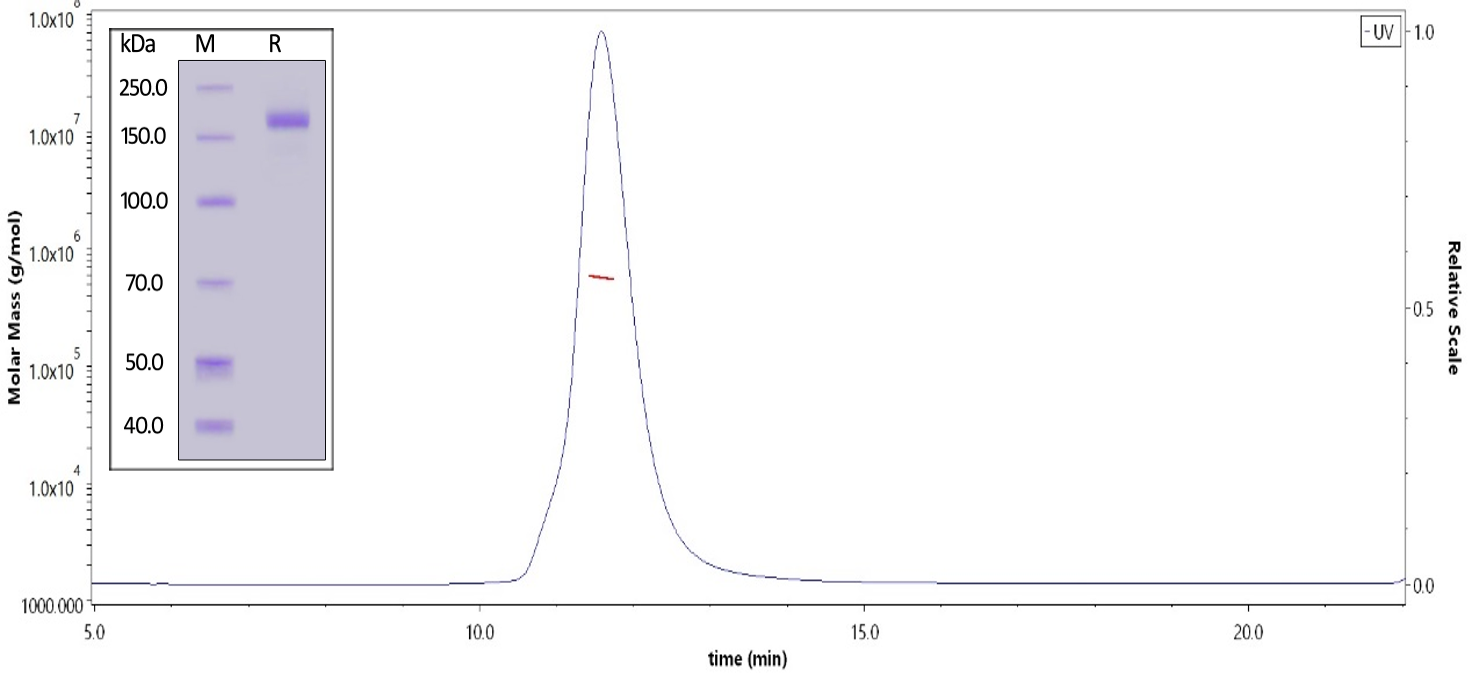
经MALS验证,抗原纯度可达95%;
3
参考多个权威数据库和最新科研进展信息,覆盖真实毒株的主要突变位点。
产品列表
| 突变种属 | 分子 | 货号 | 种属 | 标签 | 宿主 | 突变位点 | 产品描述 | 订购/预购 |
|---|---|---|---|---|---|---|---|---|
|
SARS-CoV-2 南非突变株 C.1.2 |
Spike protein | SPN-C52Hb | SARS-CoV-2 | His Tag | HEK293 | C136F, Y144del, R190S, D215G, LA242-243del, Y449H, E484K, N501Y, D614G, H655Y, N679K, T716I, T859N | SARS-CoV-2 Spike Trimer (C136F, Y144del, R190S, D215G, LA242-243del, Y449H, E484K, N501Y, D614G, H655Y, N679K, T716I, T859N), His Tag | |
| Spike protein | SPN-C82Eq | SARS-CoV-2 | His Tag & Avi Tag | HEK293 | C136F, Y144del, R190S, D215G, LA242-243del, Y449H, E484K, N501Y, D614G, H655Y, N679K, T716I, T859N | Biotinylated SARS-CoV-2 Spike Trimer (C136F, Y144del, R190S, D215G, LA242-243del, Y449H, E484K, N501Y, D614G, H655Y, N679K, T716I, T859N), His,Avitag™ | ||
| Spike RBD | SPD-C5228 | SARS-CoV-2 | His Tag | HEK293 | Y449H, E484K, N501Y | SARS-CoV-2 Spike RBD (Y449H, E484K, N501Y), His Tag | ||
| Spike RBD | SPD-C82En | SARS-CoV-2 | His Tag & Avi Tag | HEK293 | Y449H, E484K, N501Y | Biotinylated SARS-CoV-2 Spike RBD (Y449H, E484K, N501Y), His,Avitag™ | ||
| Spike NTD | SPD-C5229 | SARS-CoV-2 | His Tag | HEK293 | C136F, Y144del, R190S, D215G, LA242-243del | SARS-CoV-2 Spike NTD (C136F, Y144del, R190S, D215G, LA242-243del), His Tag | ||
|
SARS-CoV-2 哥伦比亚突变株 Mu | B.1.621 (Columbia) |
Spike protein | SPN-C52Ha | SARS-CoV-2 | His Tag | HEK293 | T95I, Y144S, Y145N, R346K, E484K, N501Y, D614G, P681H, D950N | SARS-CoV-2 Spike Trimer (T95I, Y144S, Y145N, R346K, E484K, N501Y, D614G, P681H, D950N), His Tag | |
| Spike protein | SPN-C82Ek | SARS-CoV-2 | His Tag & Avi Tag | HEK293 | T95I, Y144S, Y145N, R346K, E484K, N501Y, D614G, P681H, D950N | Biotinylated SARS-CoV-2 Spike Trimer (T95I, Y144S, Y145N, R346K, E484K, N501Y, D614G, P681H, D950N), His,Avitag™ | ||
| Spike RBD | SPD-C5221 | SARS-CoV-2 | His Tag | HEK293 | R346K, E484K, N501Y | SARS-CoV-2 Spike RBD (R346K, E484K, N501Y), His Tag | ||
| Spike RBD | SPD-C82Em | SARS-CoV-2 | His Tag & Avi Tag | HEK293 | R346K, E484K, N501Y | Biotinylated SARS-CoV-2 Spike RBD (R346K, E484K, N501Y), His,Avitag™ | ||
| Spike NTD | SPD-C5222 | SARS-CoV-2 | His Tag | HEK293 | T95I, Y144S, Y145N | SARS-CoV-2 Spike NTD (T95I, Y144S, Y145N), His Tag |
参考文献
1. Keiya Uriu, Izumi Kimura, Kotaro Shirakawa, et al. Ineffective neutralization of the SARS-CoV-2 Mu variant by convalescent and vaccine sera. The Genotype to Phenotype Japan (G2P-Japan) Consortium, So Nakagawa, Kei Sato. bioRxiv 2021.09.06.459005; doi: https://doi.org/10.1101/2021.09.06.459005
2. Katherine Laiton-Donato, Carlos Franco-Muñoz, Diego A. Álvarez-Díaz, et al. Characterization of the emerging B.1.621 variant of interest of SARS-CoV-2. medRxiv 2021.05.08.21256619; doi: https://doi.org/10.1101/2021.05.08.21256619
3. Cathrine Scheepers, Josie Everatt, Daniel G. Amoako, et al. Emergence and phenotypic characterization of C.1.2, a globally detected lineage that rapidly accumulated mutations of concern. medRxiv 2021.08.20.21262342; doi: https://doi.org/10.1101/2021.08.20.21262342


ACROBiosystems百普赛斯生物科技股份有限公司
北京:010-53681107
上海:021-50850665
邮箱:(产品订购)order.cn@acrobiosystems.com
邮箱:(技术支持)tech.cn@acrobiosystems.com
邮箱:(廉洁合规)lianjie@acrobiosystems.com
地址:北京经济技术开发区宏达北路8号5号楼4层
访问验证
为了更好的访问体验,请进行如下验证